1. Un aperçu du shell Unix 🖵#
Un shell est un programme qui attend un ordre de la part de l’utilisateur, exécute cet ordre, affiche le résultat puis attend à nouveau un ordre. En anglais on parle de REPL pour Read Execute Print Loop. Il existe plusieurs shells : Bash, csh, zsh… Ici on ne parlera que du shell Bash qui est le plus utilisé. Cela dit, toutes les commandes présentées ici sont communes à tous les shells Unix.
Lorsqu’on donne des ordres à un shell, on utilise ni menu, ni bouton, ni case à cocher. Tous les ordres sont écrits au shell.
Le shell fonctionne sur un ordinateur, qui n’a aucune capacité d’abstraction ni d’intuition. Pour que les ordres donnés au shell soient compris et exécutés, il faut respecter des règles bien précises :
Respecter la casse, c’est-à-dire l’utilisation des majuscules et des minuscules. Par exemple la commande
lsexiste mais pasLSouLs.Comprendre que le caractère espace est utilisé pour séparer l’instruction des options et des arguments et que par conséquent ce caractère ne doit plus être utilisé dans les noms de fichiers ou de répertoires.
Enfin, un terminal (ou une console) est un logiciel graphique qui lance un shell. C’est ce dernier (le shell) qui va exécuter vos instructions.
1.1. Invite de commande#
Lorsque vous lancez un shell, l’invite de commande, c’est-à-dire la zone à gauche du curseur, ressemble à quelque chose du type :
pierre@jeera:~$
ou bien encore
[ppoulain@cpu-node-16 ~]$
voire
toto $
Par convention, l’invite de commande sera représentée dans la suite de ce document par le caractère dollar «$» en tout début de ligne :
$
Pour reproduire les commandes présentées, il ne faut pas taper ce caractère en début de ligne. Ainsi,
$ pwd
signifie : tapez l’instruction pwd dans le shell (sans le $), puis validez cette commande en pressant la touche Entrée.
1.2. Préparer les données#
Pour reproduire les exemples suivants, vous allez préparer les données nécessaires. Déplacez-vous tout d’abord dans votre répertoire utilisateur avec la commande cd :
$ cd
Note
Ne tapez pas le caractère $ en début de ligne et faites attention aux majuscules et au minuscules.
puis exécutez les commandes suivant :
$ wget https://github.com/pierrepo/unix-tutorial/raw/master/content/tuto1/unix.tgz
$ tar zxvf unix.tgz
$ cd unix
Ces commandes seront en partie expliquées plus loin. Pour ne pas réécrire complètement ces commandes, vous pouvez les copier / coller avec les raccourcis Ctrl+C pour copier et Ctrl+V (ou parfois Ctrl+Maj+C) pour coller.
1.3. Parcourir les répertoires et les fichiers#
1.3.1. Savoir où on se trouve : pwd#
La première commande à connaître est la commande pwd qui signifie print working directory et qui affiche le nom du répertoire courant.
Par exemple :
$ pwd
/shared/home/ppoulain/unix
Cela signifie qu’on se trouve actuellement dans le répertoire /shared/home/ppoulain/unix.
Dans votre session, ppoulain sera bien sur remplacé par votre nom d’utilisateur sur le cluster IFB.
Sous Unix, les répertoires et les fichiers sont organisés sous forme d’une structure en arbre. On parle d’arborescence (voir l’illustration de Software Carpentry).
Le répertoire dont dépendent tous les autres est le / qu’on appelle la « racine » (root en anglais), les différents sous-répertoires sont séparés les uns des autres par le caractère / (le même caractère que la racine).
Dans le cas de /shared/home/ppoulain/unix :
on se trouve dans le répertoire
unix,qui est lui-même un sous-répertoire du répertoire
ppoulain,qui est lui-même un sous-répertoire du répertoire
home,qui est lui-même un sous-répertoire du répertoire
shared,qui est lui-même un sous-répertoire du répertoire
/(la racine du système de fichiers).
/shared/home/ppoulain/unix est aussi appelé un « chemin » car il indique la succession des répertoires à suivre pour arriver jusqu’au dernier répertoire unix. D’abord la racine /, puis shared, puis home, puis ppoulain et enfin unix.
Warning
Ne confondez pas « / » qui, tout au début d’un chemin, désigne la racine, du caractère « / » qui sépare deux répertoires successifs.
Lorsqu’un chemin débute par / (la racine), on parle de chemin absolu. Il existe aussi des chemins relatifs (donc qui ne débutent pas par /) que l’on verra plus tard.
1.3.2. Afficher le contenu d’un répertoire : ls#
La commande ls (pour list) affiche le contenu d’un répertoire :
$ ls
genomes paper.pdf protein.txt shopping.txt transferrin.csv transferrin.tsv
On peut modifier le comportement par défaut d’une commande avec une (ou plusieurs) option(s). Cette option est séparée de la commande ls par un ou plusieurs espaces.
Par exemple avec l’option -F :
$ ls -F
genomes/ paper.pdf protein.txt shopping.txt transferrin.csv transferrin.tsv
Le caractère / a été ajouté à la fin de genomes pour indiquer qu’il s’agit d’un répertoire.
Remarquez que les commandes suivantes donneraient aussi le même résultats :
$ ls -F
genomes/ paper.pdf protein.txt shopping.txt transferrin.csv transferrin.tsv
et pourquoi pas :
$ ls -F
genomes/ paper.pdf protein.txt shopping.txt transferrin.csv transferrin.tsv
Retenez qu’il faut au moins un espace entre une commande et son option.
Une autre option sympa est l’option -l (la lettre l en minuscule) qui affiche des informations complémentaires sur le contenu d’un répertoire :
$ ls -l
total 120
drwxr-xr-x 2 ppoulain ppoulain 4096 Mar 13 2019 genomes
-rw-r--r-- 1 ppoulain ppoulain 97830 Mar 13 2019 paper.pdf
-rw-r--r-- 1 ppoulain ppoulain 144 Mar 13 2019 protein.txt
-rw-r--r-- 1 ppoulain ppoulain 46 Mar 12 14:19 shopping.txt
-rw-r--r-- 1 ppoulain ppoulain 940 Mar 13 2019 transferrin.csv
-rw-r--r-- 1 ppoulain ppoulain 940 Mar 12 2019 transferrin.tsv
L’option -l peut être associée à l’option -h pour afficher des tailles lisibles par des humains, c’est-à-dire en octets, ko, Mo et Go.
$ ls -l -h
total 120K
drwxr-xr-x 2 ppoulain ppoulain 4.0K Mar 13 2019 genomes
-rw-r--r-- 1 ppoulain ppoulain 96K Mar 13 2019 paper.pdf
-rw-r--r-- 1 ppoulain ppoulain 144 Mar 13 2019 protein.txt
-rw-r--r-- 1 ppoulain ppoulain 46 Mar 12 14:19 shopping.txt
-rw-r--r-- 1 ppoulain ppoulain 940 Mar 13 2019 transferrin.csv
-rw-r--r-- 1 ppoulain ppoulain 940 Mar 12 2019 transferrin.tsv
Le fichier paper.pdf a une taille de 96 kilo octets.
Les options peuvent aussi être combinées :
$ ls -lh
total 120K
drwxr-xr-x 2 ppoulain ppoulain 4.0K Mar 13 2019 genomes
-rw-r--r-- 1 ppoulain ppoulain 96K Mar 13 2019 paper.pdf
-rw-r--r-- 1 ppoulain ppoulain 144 Mar 13 2019 protein.txt
-rw-r--r-- 1 ppoulain ppoulain 46 Mar 12 14:19 shopping.txt
-rw-r--r-- 1 ppoulain ppoulain 940 Mar 13 2019 transferrin.csv
-rw-r--r-- 1 ppoulain ppoulain 940 Mar 12 2019 transferrin.tsv
Une combinaison d’options intéressantes est -lhrt qui affiche en dernier (en bas de la liste) le fichier modifié le plus récemment :
$ ls -lhrt
total 120K
-rw-r--r-- 1 ppoulain ppoulain 940 Mar 12 2019 transferrin.tsv
drwxr-xr-x 2 ppoulain ppoulain 4.0K Mar 13 2019 genomes
-rw-r--r-- 1 ppoulain ppoulain 940 Mar 13 2019 transferrin.csv
-rw-r--r-- 1 ppoulain ppoulain 96K Mar 13 2019 paper.pdf
-rw-r--r-- 1 ppoulain ppoulain 144 Mar 13 2019 protein.txt
-rw-r--r-- 1 ppoulain ppoulain 46 Mar 12 14:19 shopping.txt
L’option -t affiche les fichiers du plus récent au plus ancien et l’option -r inverse cet ordre. Ici, le dernier fichier modifié est shopping.txt.
L’option -a affiche tout le contenu du répertoire courant, notamment les fichiers et répertoires cachés qui commencent (sous Unix) par le caractère . :
$ ls -a
. .. genomes paper.pdf protein.txt shopping.txt transferrin.csv transferrin.tsv
Par défaut, il y a deux répertoires cachés qui sont toujours présents et sont créés automatiquement : . et ..
Le répertoire
.désigne le répertoire courant.Le répertoire
..désigne le répertoire parent. Par exemple, si on est dans le répertoire/shared/home/ppoulain/unix, alors..désigne le répertoire/shared/home/ppoulain.
Enfin, la commande ls peut aussi afficher le contenu d’un répertoire passé en argument :
$ ls genomes
NC_000907_head.gbk NC_004459_head.gbk NC_007168_head.gbk NC_011333_head.gbk
NC_000964_head.gbk NC_004461_head.gbk NC_007350_head.gbk NC_011852_head.gbk
NC_002505_head.gbk NC_004917_head.gbk NC_009033_head.gbk NC_012655_head.gbk
NC_002570_head.gbk NC_006298_head.gbk NC_009477_head.gbk NC_013893_head.gbk
NC_002976_head.gbk NC_006840_head.gbk NC_011184_head.gbk NC_014205_head.gbk
Comme pour les options, il faut au moins un espace entre une commande et son ou ses arguments.
On peut même obtenir des informations détaillées sur un fichier en particulier :
$ ls -lh paper.pdf
-rw-r--r-- 1 ppoulain ppoulain 96K Mar 13 2019 paper.pdf
Dans cet exemple, on apprend que le fichier paper.pdf appartient à l’utilisateur ppoulain, qu’il a été modifié pour la dernière fois le 13 mars 2019 et qu’il a une taille de 96 kilo-octets.
Note
Dans cet exemple, ls est la commande Unix, -lh sont les options et paper.pdf est l’argument.
Cette anatomie d’une instruction Unix est schématisée dans l’illustration suivante :
Source : Software Carpentry.
1.3.3. Se documenter : man et --help#
La commande man (pour manuel) affiche la documentation associée à une commande. Par exemple pour la commande ls :
$ man ls
Utilisez les flèches ↓ et ↑ pour naviguer dans cette aide. La touche Espace saute une page à la fois. La touche Q quitte l’aide et revient au shell.
Par ailleurs, de nombreuses commandes Unix disposent de l’option --help qui affiche une aide succincte :
$ ls --help
Usage: ls [OPTION]... [FILE]...
List information about the FILEs (the current directory by default).
Sort entries alphabetically if none of -cftuvSUX nor --sort is specified.
Mandatory arguments to long options are mandatory for short options too.
-a, --all do not ignore entries starting with .
-A, --almost-all do not list implied . and ..
--author with -l, print the author of each file
-b, --escape print C-style escapes for nongraphic characters
--block-size=SIZE scale sizes by SIZE before printing them; e.g.,
[...]
1.3.4. Prêter attention aux noms de fichiers et répertoires#
Sous Unix, les noms de fichiers et de répertoires sont sensibles à la casse, c’est-à-dire aux majuscules et aux minuscules. Par conséquent, le shell fait la différence entre le fichier test.txt et le fichier Test.txt.
Par ailleurs, l’extension de fichier est facultative et purement indicative. Le shell ne l’utilise pas. Elle sert simplement à l’utilisateur pour mieux organiser ses fichiers. Un nom de fichier du type test ou test.my_extension est parfaitement valide. On peut même nommer un répertoire directory.txt (mais ce n’est pas très malin 🙄).
Enfin, pour nommer un fichier ou un répertoire, on peut utiliser :
les lettres minuscules,
les lettres majuscules,
les chiffres,
le tiret du milieu «
-» (mais pas comme premier caractère du nom),le tiret du bas «
_»,le point «
.».
On vous déconseille fortement l’utilisation d’autres caractères comme +()[]éèàùç. Certains caractères sont même interdits (<>|;&).
Enfin, ne mettez jamais un espace dans un nom de fichier ou de répertoire. J-A-M-A-I-S. Unix l’autorise mais c’est très pénible par la suite car l’espace est utilisé pour séparer les options et les arguments d’une commande.
Pour le reste, laisser faire votre imagination et utilisez des noms de fichiers et de répertoires expressifs.
1.3.5. Se déplacer : cd#
La commande cd (pour change directory) permet de se déplacer d’un répertoire à l’autre. Par exemple :
$ pwd
/shared/home/ppoulain/unix
$ cd genomes
$ pwd
/shared/home/ppoulain/unix/genomes
Partant du répertoire /shared/home/ppoulain/unix, on s’est déplacé dans le répertoire /shared/home/ppoulain/unix/genomes.
Dans l’instruction cd genomes, le répertoire genomes est un argument de la commande cd. Ici, le répertoire genomes est un chemin relatif (car il ne débute pas par /). C’est-à-dire qu’on a indiqué le nom de ce répertoire (genomes) par rapport au répertoire dans lequel nous étions (/shared/home/ppoulain/unix). Bien sûr, la commande cd fonctionne très bien avec un chemin absolu. La commande
$ cd /shared/home/ppoulain/unix/genomes
conduirait au même résultat.
Un moyen simple de revenir dans le répertoire parent est d’utiliser le raccourci .. :
$ pwd
/shared/home/ppoulain/unix/genomes
$ cd ..
$ pwd
/shared/home/ppoulain/unix
Un autre raccourci pratique pour revenir dans le répertoire utilisateur (répertoire par défaut dans lequel se trouve l’utilisateur lorsqu’il lance un shell) est ~ :
$ pwd
/shared/home/ppoulain/unix
$ cd ~
$ pwd
/shared/home/ppoulain
Note
Simplement taper la commande cd (sans argument) ramène aussi l’utilisateur dans son répertoire personnel.
1.3.6. Créer un répertoire : mkdir#
Revenez au préalable dans le bon répertoire avec la commande :
$ cd ~/unix
$ pwd
/shared/home/ppoulain/unix
La commande mkdir (pour make directory) crée un répertoire :
$ ls
genomes paper.pdf protein.txt shopping.txt transferrin.csv transferrin.tsv
$ mkdir test
$ ls
genomes paper.pdf protein.txt shopping.txt test transferrin.csv transferrin.tsv
1.3.7. Copier : cp#
La commande cp (pour copy) copie un fichier vers un nouveau fichier :
$ ls
genomes paper.pdf protein.txt shopping.txt test transferrin.csv transferrin.tsv
$ cp paper.pdf article.pdf
$ ls
article.pdf genomes paper.pdf protein.txt shopping.txt test transferrin.csv transferrin.tsv
cp peut copier plusieurs fichiers dans un répertoire :
$ cp paper.pdf article.pdf test
$ ls test
article.pdf paper.pdf
C’est toujours le dernier argument de la ligne de commande qui est utilisé comme destination (ici test) de la copie.
cp peut aussi copier un répertoire dans un autre répertoire avec l’option -r :
$ cp -r genomes test
$ ls test
article.pdf genomes paper.pdf
1.3.8. Visualiser un bout d’arborescence : tree#
La commande tree permet de visualiser une partie de l’arborescence des répertoires et des fichiers. Par exemple, pour visualiser l’arborescence du répertoire test :
$ tree test/
test/
├── article.pdf
├── genomes
│ ├── NC_000907_head.gbk
│ ├── NC_000964_head.gbk
│ ├── NC_002505_head.gbk
│ ├── NC_002570_head.gbk
│ ├── NC_002976_head.gbk
│ ├── NC_004459_head.gbk
│ ├── NC_004461_head.gbk
│ ├── NC_004917_head.gbk
│ ├── NC_006298_head.gbk
│ ├── NC_006840_head.gbk
│ ├── NC_007168_head.gbk
│ ├── NC_007350_head.gbk
│ ├── NC_009033_head.gbk
│ ├── NC_009477_head.gbk
│ ├── NC_011184_head.gbk
│ ├── NC_011333_head.gbk
│ ├── NC_011852_head.gbk
│ ├── NC_012655_head.gbk
│ ├── NC_013893_head.gbk
│ └── NC_014205_head.gbk
└── paper.pdf
1 directory, 22 files
1.3.9. Renommer et déplacer : mv#
La commande mv (pour move) renomme des fichiers ou des répertoires :
$ ls
article.pdf genomes paper.pdf protein.txt shopping.txt test transferrin.csv transferrin.tsv
$ mv article.pdf article2.pdf
$ ls
article2.pdf genomes paper.pdf protein.txt shopping.txt test transferrin.csv transferrin.tsv
$ mv test test2
$ ls
article2.pdf genomes paper.pdf protein.txt shopping.txt test2 transferrin.csv transferrin.tsv
mv déplace aussi des fichiers ou des répertoires dans un autre répertoire :
$ ls
article2.pdf genomes paper.pdf protein.txt shopping.txt test2 transferrin.csv transferrin.tsv
$ mkdir test3
$ mv article2.pdf test2 test3
$ ls test3
article2.pdf test2
$ ls
genomes paper.pdf protein.txt shopping.txt test3 transferrin.csv transferrin.tsv
1.3.10. Supprimer : rm#
La commande rm (pour remove) supprime des fichiers ou des répertoires :
$ cd test3
$ ls
article2.pdf test2
$ rm article2.pdf
$ ls
test2
Pour que rm réalise la suppression sans demander de confirmation, il faut utiliser l’option-f.
Pour supprimer un répertoire, il faut utiliser l’option supplémentaire -r :
$ rm -rf test2
$ ls
Warning
Il n’y a pas de corbeille dans le shell Unix, c’est-à-dire qu’il n’y a aucune possibilité de récupérer un fichier ou un répertoire effacé par erreur. Pensez à sauvegarder régulièrement vos fichiers.
1.3.11. Automatiser (un peu)#
Les caractères * et ? peuvent remplacer n’importe quels caractères dans les noms de fichiers ou de répertoires :
*remplace 0, 1 ou plus caractères.?remplace exactement 1 caractère.
Par exemple :
$ ls *txt
protein.txt shopping.txt
$ ls transferrin.?sv
transferrin.csv transferrin.tsv
Ces caractères sont très utiles quand il s’agit d’effectuer une opération sur plusieurs fichiers en même temps :
$ mkdir test4
$ cp transferrin.* test4
$ ls test4
transferrin.csv transferrin.tsv
La commande cp transferrin.* test4 est un raccourci pour
cp transferrin.csv transferrin.tsv test4
Cela fonctionne avec autant de fichiers qu’on le souhaite.
1.4. Trucs et astuces#
Écrire des commandes dans un shell peut sembler rébarbatif mais on gagne rapidement en efficacité avec les astuces suivantes.
1.4.1. Rappel des anciennes commandes#
La flèche du haut ↑ rappelle les commandes précédentes (depuis la plus récente jusqu’à la plus ancienne).
1.4.2. Complétion automatique#
La touche tabulation Tab est utile pour compléter une commande, un nom de répertoire ou de fichier. Appuyez deux fois sur Tab en cas d’ambiguïté.
Par exemple, si vous tapez /h puis la touche Tab, le shell va compléter automatiquement à /home.
1.4.3. Arrêter une commande en cours#
Pressez la touche Ctrl et la touche C en même temps pour arrêter une commande en cours.
1.4.4. Copier / coller#
Pour copier / coller quelque chose dans le shell, sélectionnez du texte puis utilisez les combinaisons de touches Ctrl+C pour copier et Ctrl+V (ou Ctrl+Maj+V) pour coller.
1.5. Explorer le contenu de fichiers#
Jusqu’à présent, on s’est contenté d’afficher le contenu de répertoires et de déplacer ou copier des fichiers, sans connaître leur contenu.
Pour afficher le contenu d’un fichier, on utilise la commande cat (pour concatenate) :
$ cat shopping.txt
banana 21
pineaple 1
pear 3
apple 10
orange 4
Bien sûr, afficher le contenu d’un fichier dans un terminal n’a de sens ici que pour un fichier texte. En effet, afficher le contenu d’un fichier binaire produit une suite de caractères incompréhensibles.
$ cat paper.pdf
xrefbjfalse/SM 0.02/Type/ExtGState>>
0 1
0000000000 65535 f
11 2
0000073672 00000 n
0000077288 00000 n
15 1
0000077496 00000 n
48 4
0000077860 00000 n
0000097345 00000 n
0000097389 00000 n
0000097434 00000 n
trailer
<</Size 52/Root 14 0 R/Info 12 0 R/ID[<C52AD85A8BAFBD722C6FCD30421B945E><33F927FA361D6A4FA1263C03468E9074>]/Prev 116>>
startxref
97487
%%EOF
Note
Dans la suite, nous n’explorerons que le contenu de fichiers textes.
La commande cat n’est utile que si le fichier est assez court. Si ce n’est pas le cas, le contenu du fichier va défiler d’un seul coup à l’écran, sans qu’on puisse en voir le début. Par exemple :
$ cat transferrin.csv
1TFD,Oryctolagus cuniculus,304
2D3I,Gallus gallus,686
2O84,Homo sapiens,337
3FGS,Homo sapiens,337
3QYT,Homo sapiens,679
3SKP,Homo sapiens,342
4H0W,Homo sapiens,679
4X1B,Homo sapiens,679
5H52,Homo sapiens,679
5WTD,Homo sapiens,679
5X5P,Homo sapiens,679
6CTC,Homo sapiens,679
[...]
Pour les gros fichiers, on préfère utiliser la commande less qui affiche progressivement le contenu d’un fichier :
$ less transferrin.csv
Comme avec la commande man, les flèches ↓ et ↑ permettent de naviguer dans le contenu du fichier. La touche Espace saute une page à la fois. La touche Q quitte less et revient au shell.
La commande head affiche les premières lignes d’un fichier :
$ head transferrin.csv
1A8E,Homo sapiens,329
1A8F,Homo sapiens,329
1AIV,Gallus gallus,686
1AOV,Anas platyrhynchos,686
1B3E,Homo sapiens,330
1D3K,Homo sapiens,329
1D4N,Homo sapiens,329
1DOT,Anas platyrhynchos,686
1DTG,Homo sapiens,334
1FQE,Homo sapiens,331
Par défaut, head affiche les 10 premières lignes du fichier. L’option -n précise le nombre de lignes à afficher. Par exemple :
$ head -n 2 transferrin.csv
1A8E,Homo sapiens,329
1A8F,Homo sapiens,329
Réciproquement, la commande tail affiche les dernières lignes d’un fichier texte. L’option -n est également disponible :
$ tail transferrin.csv
2O84,Homo sapiens,337
3FGS,Homo sapiens,337
3QYT,Homo sapiens,679
3SKP,Homo sapiens,342
4H0W,Homo sapiens,679
4X1B,Homo sapiens,679
5H52,Homo sapiens,679
5WTD,Homo sapiens,679
5X5P,Homo sapiens,679
6CTC,Homo sapiens,679
$ tail -n 2 transferrin.csv
5X5P,Homo sapiens,679
6CTC,Homo sapiens,679
Dans l’exemple ci-dessus, on a affiché les 2 dernières lignes du fichier transferrin.csv.
1.6. Créer ou éditer un fichier texte#
Nano est un éditeur de texte qui fonctionne dans un shell, donc sans interface graphique, sans menu, sans icône, contrairement à des éditeurs de texte comme Notepad++.
Pour le lancer, on utilise la commande nano :
$ nano
Et on obtient quelque chose du type :
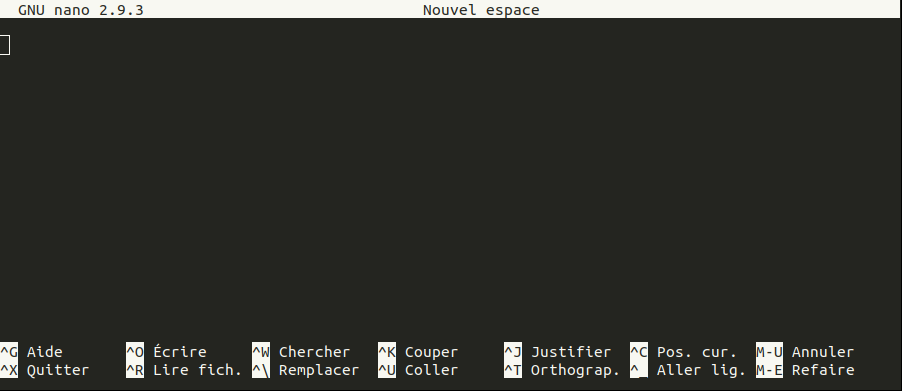
Selon la version de votre système Unix, il se peut que l’interface soit en anglais mais les raccourcis clavier seront les mêmes.
On peut tout de suite commencer à taper du texte.
Pour sauvegarder le texte ainsi entré dans nano, on utilise la combinaison de touches Ctrl+O (c’est-à-dire qu’on presse en même temps les touches Ctrl et O). On entre ensuite le nom qu’on souhaite donner au fichier (par exemple test.txt) puis on valide par la touche Entrée.
Les différentes combinaisons de touches sont rappelées en bas de l’écran. Le caractère « ^ » désigne la touche Ctrl.
On peut continuer à éditer le fichier puis l’enregistrer, et ainsi de suite.
Pour quitter nano, on utilise la combinaison de touches Ctrl+X.
On se retrouve alors dans le shell et on peut vérifier que le fichier (ici test.txt) a bien été créé dans le répertoire courant.
$ ls
genomes paper.pdf protein.txt shopping.txt test.txt transferrin.csv transferrin.tsv
On peut aussi ouvrir un fichier texte existant en indiquant en argument le nom du fichier à ouvrir :
$ nano shopping.txt
Modifiez le fichier shopping.txt avec nano :
ajouter la ligne
kiwi 16à la fin du fichier,remplacer le nombre d’oranges par
8.
Enregistrez le fichier (Ctrl+O puis Entrée) et enfin quitter nano (Ctrl+X).
Vérifiez avec la commande cat que les modifications ont bien été prises en compte :
$ cat shopping.txt
Aide
Le fichier shopping.txt devrait ressembler à ceci :
banana 21
pineaple 1
pear 3
apple 10
orange 8
kiwi 16
1.7. Manipuler des données#
1.7.1. Compter : wc#
La commande wc (pour word count) compte le nombre de lignes, de mots et de caractères d’un fichier.
$ wc shopping.txt
6 12 54 shopping.txt
On apprend ainsi que le fichier shopping.txt contient 6 lignes, 12 mots et 54 caractères, ou des valeurs équivalentes si vous avez modifé ce fichier.
L’option -l indique à la commande wc de ne compter que le nombre de lignes. Et réciproquement pour -w et le nombre de mots, et -c et le nombre de caractères.
Lorsque plusieurs fichiers sont fournis en argument à la commande wc, le total est aussi renvoyé :
$ wc -l transferrin.*
41 transferrin.csv
41 transferrin.tsv
82 total
Les fichiers transferrin.csv et transferrin.tsv contiennent chacun 41 lignes, soit un total de 82 lignes.
1.7.2. Trier : sort#
La commande sort trie le contenu d’un fichier.
$ cat shopping.txt
banana 21
pineaple 1
pear 3
apple 10
orange 8
kiwi 16
$ sort shopping.txt
apple 10
banana 21
kiwi 16
orange 8
pear 3
pineaple 1
Les lignes ont été triées par ordre alphabétique.
La commande sort a également la notion de colonnes (ou de champs). Par défaut, le séparateur de champs est un caractère blanc (espace, tabulation). Dans le fichier shopping.txt, sort trouve une première colonne avec le nom des fruits et une seconde avec les quantités.
On peut trier le fichier shopping.txt suivant le nombre de fruits en indiquant à sort d’utiliser la 2e colonne avec l’option -k :
$ sort -k 2 shopping.txt
pineaple 1
apple 10
kiwi 16
banana 21
pear 3
orange 8
Les lignes sont alors triées suivant la seconde colonne, mais par ordre alphabétique, ce qui explique que 10 soit avant 3. Pour trier explicitement des valeurs numériques, on utilise l’option -g :
$ sort -k 2 -g shopping.txt
pineaple 1
pear 3
orange 8
apple 10
kiwi 16
banana 21
L’ordre numérique est ainsi respecté puisque 3 est plus petit (donc situé avant) que 10.
Enfin l’option -r inverse le tri initial :
$ sort -r shopping.txt
pineaple 1
pear 3
orange 8
kiwi 16
banana 21
apple 10
On peut aussi utiliser l’option -r pour trier des valeurs numériques (option -g) sur une colonne donnée (option -k) :
$ sort -k 2 -g -r shopping.txt
banana 21
kiwi 16
apple 10
orange 8
pear 3
pineaple 1
1.7.3. Trouver les éléments uniques : uniq#
La commande uniq affiche les éléments uniques.
Par exemple avec le contenu du fichier protein.txt :
$ cat protein.txt
insulin
insulin
insulin
insulin
insulin
insulin
integrin
integrin
rhodopsin
rhodopsin
rhodopsin
transferrin
transferrin
transferrin
transferrin
On obtient alors uniquement les lignes uniques :
$ uniq protein.txt
insulin
integrin
rhodopsin
transferrin
L’option -c compte le nombre de fois qu’un élément est présent :
$ uniq -c protein.txt
6 insulin
2 integrin
3 rhodopsin
4 transferrin
Note
La commande uniq ne fonctionne que sur un fichier trié, c’est-à-dire pour lequel les lignes sont déjà triées par ordre alphabétique.
1.7.4. Extraire un colonne : cut#
La commande cut extrait une ou plusieurs colonnes d’un fichier.
Par exemple avec le fichier transferrin.csv qui contient les identifiants PDB de structures de transferrines, avec l’organisme d’origine et le nombre d’acides aminés de la structure :
$ head -n 5 transferrin.csv
1A8E,Homo sapiens,329
1A8F,Homo sapiens,329
1AIV,Gallus gallus,686
1AOV,Anas platyrhynchos,686
1B3E,Homo sapiens,330
On souhaite extraire la 2e colonne qui contient des noms d’organismes :
$ cut -d "," -f 2 transferrin.csv
Homo sapiens
Homo sapiens
Gallus gallus
Anas platyrhynchos
Homo sapiens
Homo sapiens
[...]
L’option -d spécifie le délimiteur, c’est-à-dire le caractère utilisé pour séparer les différents champs (les colonnes). Par défaut, cut utilise la tabulation. Nous manipulons ici un fichier au format CSV (pour comma-separated values) dont les colonnes sont séparées par une virgule.
L’option -f précise le numéro du champ qu’on souhaite extraire (ici le 2e champ).
1.8. Gérer les flux : redirection et emboîtement#
On souhaite extraire du fichier transferrin.csv la liste des différents organismes d’où proviennent les structures de transferrines. On a besoin pour cela de réaliser 3 étapes :
Extraire la liste des organismes (avec
cut).Trier par ordre alphabétique ces organismes (avec
sort).Trouver les différents organismes (avec
uniq). L’étape 2 est justifiée par le fait queuniqne fonctionne que sur des données triées.
Pour stocker l’information, d’une étape à l’autre, on peut renvoyer le résultat dans un fichier avec la redirection «>».
1.8.1. Étape 1 (cut)#
$ cut -d "," -f 2 transferrin.csv > organism.txt
Le résultat de la commande ne s’affiche pas à l’écran mais est redirigé dans le fichier organism.txt.
On peut jeter un oeil au contenu de organism.txt avec les commandes cat, less, head ou tail :
$ head organism.txt
Homo sapiens
Homo sapiens
Gallus gallus
Anas platyrhynchos
Homo sapiens
Homo sapiens
Homo sapiens
Anas platyrhynchos
Homo sapiens
Homo sapiens
1.8.2. Étape 2 (sort)#
$ sort organism.txt > organism_sorted.txt
Ici encore, rien ne s’affiche à l’écran mais on peut contrôler le résultat :
$ head organism_sorted.txt
Anas platyrhynchos
Anas platyrhynchos
Gallus gallus
Gallus gallus
Gallus gallus
Gallus gallus
Gallus gallus
Gallus gallus
Gallus gallus
1.8.3. Étape 3 (uniq)#
$ uniq organism_sorted.txt
Anas platyrhynchos
Gallus gallus
Homo sapiens
Oryctolagus cuniculus
Sus scrofa
Les structures de transferrines du fichier transferrin.csv proviennent de 5 organismes différents.
1.8.4. Emboîtement#
Pour réaliser l’analyse précédente, nous avons dû créer deux fichiers intermédiaires : organism.txt et organism_sorted.txt.
Pour éviter la création de ces fichiers temporaires et réaliser l’analyse en une seule fois, on emboîte (« chaîne ») les différentes étapes. La sortie produite par une étape devient l’entrée de l’étape suivante (voir l’illustration de Software Carpentry). On utilise pour cela le caractère «|» :
$ cut -d "," -f 2 transferrin.csv | sort | uniq
Anas platyrhynchos
Gallus gallus
Homo sapiens
Oryctolagus cuniculus
Sus scrofa
On obtient le même résultat que précédemment mais en une seule étape et sans fichier intermédiaire.
Si on souhaite obtenir le nombre de structures par organisme, on peut très rapidement modifier la commande précédente en utilisant l’option -c de uniq :
$ cut -d "," -f 2 transferrin.csv | sort | uniq -c
2 Anas platyrhynchos
10 Gallus gallus
26 Homo sapiens
2 Oryctolagus cuniculus
1 Sus scrofa
Et si on préfère travailler avec le fichier transferrin.tsv dont les colonnes sont séparées par des tabulations :
$ cut -f 2 transferrin.tsv | sort | uniq -c
2 Anas platyrhynchos
10 Gallus gallus
26 Homo sapiens
2 Oryctolagus cuniculus
1 Sus scrofa
Rappel
La tabulation est le caractère par défaut utilisé par la commande cut pour séparer les différentes colonnes.
1.9. Chercher#
1.9.1. Chercher dans des fichiers : grep#
La commande grep cherche un motif dans un ou plusieurs fichiers et renvoie les lignes correspondantes :
$ grep "apple" shopping.txt
apple 10
Le motif à rechercher est indiqué entre guillemets.
L’option -n affiche le numéro de la ligne dans lequel est trouvé le motif :
$ grep -n "apple" shopping.txt
4:apple 10
Si on recherche un motif dans plusieurs fichiers, le nom des fichiers dans lequel le motif est trouvé est également affiché :
$ grep "DEFI" genomes/*.gbk
genomes/NC_000907_head.gbk:DEFINITION Haemophilus influenzae Rd KW20, complete genome.
genomes/NC_000964_head.gbk:DEFINITION Bacillus subtilis subsp. subtilis str. 168, complete genome.
genomes/NC_002505_head.gbk:DEFINITION Vibrio cholerae O1 biovar El Tor str. N16961 chromosome I, complete
genomes/NC_002570_head.gbk:DEFINITION Bacillus halodurans C-125, complete genome.
genomes/NC_002976_head.gbk:DEFINITION Staphylococcus epidermidis RP62A, complete genome.
genomes/NC_004459_head.gbk:DEFINITION Vibrio vulnificus CMCP6 chromosome I, complete genome.
genomes/NC_004461_head.gbk:DEFINITION Staphylococcus epidermidis ATCC 12228, complete genome.
genomes/NC_004917_head.gbk:DEFINITION Helicobacter hepaticus ATCC 51449, complete genome.
genomes/NC_006298_head.gbk:DEFINITION Haemophilus somnus 129PT plasmid pHS129, complete sequence.
genomes/NC_006840_head.gbk:DEFINITION Vibrio fischeri ES114 chromosome I, complete genome.
genomes/NC_007168_head.gbk:DEFINITION Staphylococcus haemolyticus JCSC1435, complete genome.
genomes/NC_007350_head.gbk:DEFINITION Staphylococcus saprophyticus subsp. saprophyticus ATCC 15305,
genomes/NC_009033_head.gbk:DEFINITION Staphylothermus marinus F1 chromosome, complete genome.
genomes/NC_009477_head.gbk:DEFINITION Staphylococcus aureus subsp. aureus JH9 plasmid pSJH901, complete
genomes/NC_011184_head.gbk:DEFINITION Vibrio fischeri MJ11 chromosome I, complete sequence.
genomes/NC_011333_head.gbk:DEFINITION Helicobacter pylori G27, complete genome.
genomes/NC_011852_head.gbk:DEFINITION Haemophilus parasuis SH0165, complete genome.
genomes/NC_012655_head.gbk:DEFINITION Bacillus anthracis str. A0248 plasmid pXO2, complete sequence.
genomes/NC_013893_head.gbk:DEFINITION Staphylococcus lugdunensis HKU09-01 chromosome, complete genome.
genomes/NC_014205_head.gbk:DEFINITION Staphylothermus hellenicus DSM 12710 chromosome, complete genome.
On peut bien sûr emboîter / chaîner les commandes grep si on cherche plusieurs motifs :
$ grep "DEFI" genomes/*.gbk | grep "Staphyloco"
genomes/NC_002976_head.gbk:DEFINITION Staphylococcus epidermidis RP62A, complete genome.
genomes/NC_004461_head.gbk:DEFINITION Staphylococcus epidermidis ATCC 12228, complete genome.
genomes/NC_007168_head.gbk:DEFINITION Staphylococcus haemolyticus JCSC1435, complete genome.
genomes/NC_007350_head.gbk:DEFINITION Staphylococcus saprophyticus subsp. saprophyticus ATCC 15305,
genomes/NC_009477_head.gbk:DEFINITION Staphylococcus aureus subsp. aureus JH9 plasmid pSJH901, complete
genomes/NC_013893_head.gbk:DEFINITION Staphylococcus lugdunensis HKU09-01 chromosome, complete genome.
L’option -c de grep indique combien de génomes de staphylocoques ont été trouvés :
$ grep "DEFI" genomes/*.gbk | grep -c "Staphyloco"
6
1.9.2. Petit défi#
Construisez l’enchaînement de commandes Unix qui :
cherche le motif
DEFINITIONdans tous les fichiers GenBank du répertoiregenomes;cherche les génomes de staphylocoques (
Staphylococcus) ;ne conserve que les noms des organismes ;
renvoie le tout dans le fichier
all_staphylo.txt.
Solution
Voici une solution possible :
$ grep "DEFI" genomes/*.gbk | grep "Staphylo" | cut -d " " -f 3- > all_staphylo.txt
1.9.3. Chercher des fichiers : find#
La commande find recherche des fichiers ou des répertoires.
On revient tout d’abord dans le répertoire parent :
$ pwd
/shared/home/ppoulain/unix
$ cd ..
$ pwd
/shared/home/ppoulain
On cherche maintenant les fichiers avec l’extension .csv :
$ find ./ -name "*.csv"
./unix/transferrin.csv
La commande find prend comme argument l’endroit à partir duquel on cherche, ici le répertoire courant désigné par ./ (on aurait aussi pu simplement indiquer « . »). Puis on précise le critère de recherche avec l’option -name, ici tous les fichiers qui se terminent par .csv.
1.10. Se souvenir#
La commande history affiche toutes les commandes que vous avez entrées, de la plus ancienne à la plus récente.
Ainsi, la commande
$ history
produit une sortie du type :
[...]
582 grep "apple" shopping.txt
583 grep -n "apple" shopping.txt
584 grep "DEFI" genomes/*.gbk
585 grep "DEFI" genomes/*.gbk | grep "Staphyloco"
586 grep "DEFI" genomes/*.gbk | grep -c "Staphyloco"
587 pwd
588 cd ..
589 pwd
590 find ./ -name "*.csv"
591 history
Le numéro à côté de chaque commande est le numéro de la commande dans l’ordre où elle a été lancée dans le shell. Vous aurez bien sûr des numéros et des commandes différentes.
On peut très simplement rappeler une commande à partir de son numéro précédé du caractère «!». Par exemple :
$ !590
find ./ -name "*.csv"
./unix/transferrin.csv
Enfin, lorsqu’on a travaillé un moment dans le shell, il peut être utile de sauvegarder l’historique des commandes qu’on a lancées avec :
$ history > 2019-03-14_history.txt
Ici, 2019-03-14 représente la date au format ISO 8601 AAAA-MM-JJ (année-mois-jour).
Pour automatiser cette sauvegarde avec la date du jour :
$ history > $(date --iso-8601)_history.txt
Nous verrons le fonctionnement de $(date --iso-8601) prochainement.
1.11. Préparation de la prochaine séance#
Afin de vérifier que vous avez les droits nécessaires pour réaliser les activités pratiques de la prochaine séance, exécutez dans un shell la commande suivante :
$ touch /shared/projects/2501_duo/$USER/test
Signalez tout problème ou message d’erreur.
